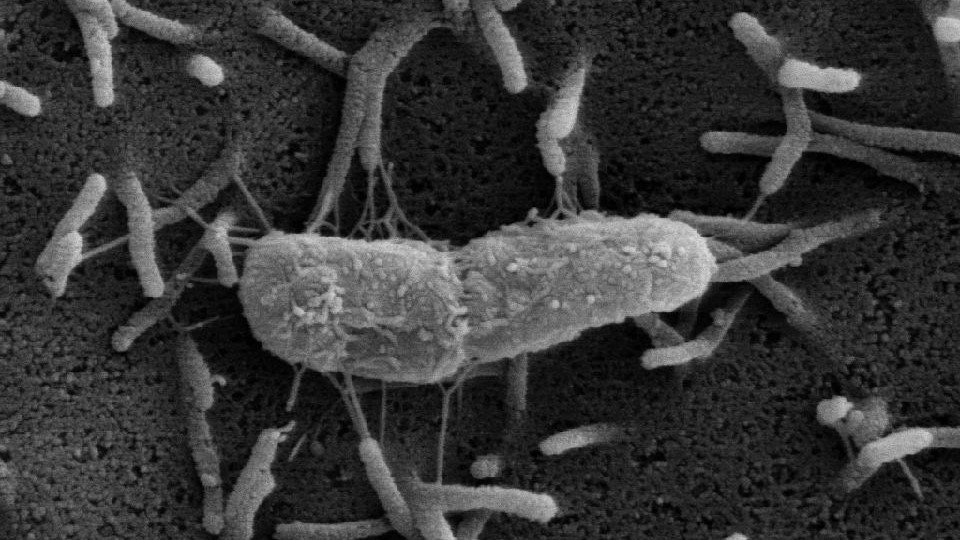
Hierbei handelt es sich um ein bewegliches genetisches Element, das die Resistenz gegen bestimmte Antibiotika auf unterschiedliche Bakterien überträgt. Dieser Mechanismus kann das Risiko für schwer einzudämmende Infektionsausbrüche in Krankenhäusern erhöhen.
Multiresistente Bakterien, gegen die verschiedenste Antibiotika machtlos sind, stellen Ärzte und Gesundheitsbehörden in Deutschland immer häufiger vor kaum lösbare Probleme. Für Krankenhausinfektionen spielen neben den bekannten Staphylococcus-aureus-Stämmen zunehmend multiresistente gramnegative Bakterien – Enterobacteriaceen – eine wichtige Rolle. Bei einem aktuellen Ausbruch in einem hessischen Krankenhaus konnten DZIF-Wissenschaftler aus Gießen gehäuft verschiedene gramnegative Bakterien nachweisen, die gegen Carbapenem resistent waren. Carbapeneme sind breit wirksame Antibiotika, die als Reserveantibiotika in Notfällen zum Einsatz kommen. Die Forscher identifizierten bei diesen Stämmen ein bewegliches genetisches Element, ein Plasmid, das für die Resistenz verantwortlich ist. Dieses neu entdeckte Multiresistenzplasmid kann auf unterschiedliche Keime übertragen werden und stellt somit eine ganz neue Dimension für Ausbrüche dar. Bei einer Infektion mit neu auftretenden Carbapenem-resistenten Erregern kann eine ausweglose Situation entstehen, in der es keine Behandlungsoption mehr gibt.
„Genau an diesem Punkt setzen die Aktivitäten der Forschungseinheit „Krankenhauskeime und Antibiotikaresistente Bakterien“ des DZIF an“, erklärt Prof. Dr. Trinad Chakraborty, Co-Koordinator der Forschungseinheit und Direktor des Instituts für Medizinische Mikrobiologie in Gießen. Dabei konzentrieren sich die Arbeiten im DZIF auf ein verbessertes Hygienemanagement, um die Weiterverbreitung multiresistenter gramnegativer Bakterien einzudämmen und Strategien zur Bekämpfung neu auftretender Infektionen und Resistenzen zu entwickeln.
Die DZIF-Bioinformatik am Standort Gießen ist in der Lage, Genomsequenzierungs-Daten zu analysieren und schnell und präzise die verantwortlichen Resistenzgene, die möglichen Übertragungsmechanismen und die Ausbruchsstämme zu identifizieren. Im Mittelpunkt steht dabei die Untersuchung beweglicher genetischer Elemente, zum Beispiel von Plasmiden.
Bei dem aktuellen Ausbruch 2014 in Hessen konnten die Wissenschaftler gehäuft verschiedene gramnegative Bakterien (Citrobacter freundii, Enterobacter aerogenes, Escherichia coli, Klebsiella oxytoca u.a.) nachweisen, die aufgrund ihrer Resistenz gegen Carbapeneme (KPC‑2) als sogenannte 4MRGN-Erreger klassifiziert wurden. Dazu zählen die multiresistenten gramnegativen Bakterien mit Resistenzen gegen Acylureidopencilline, Cephalosporine der 3. und 4. Generation, Carbapeneme und Fluorchinolone.
Die in diesem Fall ungewöhnlich breite Speziesverteilung bei gleichem Resistenzmuster machte die Anwesenheit eines extrem beweglichen genetischen Resistenzelementes wahrscheinlich. Zur Überprüfung wurden 21 repräsentative KPC-2-positive Stämme in der Medizinischen Mikrobiologie der JLU Gießen sequenziert. Die anschließende bioinformatorische Auswertung der Daten konnte das blaKPC-2-Gen auf einem bestimmten Plasmid lokalisieren, das bei jedem der untersuchten Isolate vorhanden war. Dies beweist, dass die Häufung der Carbapenemase-exprimierenden Bakterien kein Zufall war, sondern durch die rasante Ausbreitung eines spezifischen Resistenz-Plasmids verursacht wurde. Diese Untersuchung beschreibt weltweit erstmals den nosokomialen – im Krankenhaus erworbenen – Ausbruch eines Carbapenemase tragenden beweglichen genetischen Elements. Die Carbapenemase ist ein Enzym, das Carbapeneme unwirksam macht und damit Resistenz gegen dieses Antibiotikum verleiht. Ein Multiresistenzplasmid, das auf unterschiedliche Keime übertragen werden kann, tritt somit an die Stelle der Verbreitung eines klassischen resistenten Krankenhauskeims.
Besonders gefährlich kann es werden, wenn ein solches Plasmid auf pathogene Keime wie Escherichia coli oder Klebsiella pneumoniae übertragen wird. Die Infektion mit einer Kombination aus krankmachendem Erreger und Antibiotikaresistenz kann möglicherweise nicht mehr therapiert werden. Eine weitere kaum abschätzbare Bedrohung entsteht, wenn ein genesener Patient die multiresistenten Erreger mit nach Hause nimmt. Das Risiko für seine Umgebung, Familie und Kollegen ist kaum abschätzbar.
Die im Institut für Medizinischen Mikrobiologie in Gießen vorhandene Infrastruktur sowie die erfolgreiche Zusammenarbeit mit den Landes- und Bundesbehörden, dem Nationalen Referenzzentrum für gramnegative Krankenhauserreger und dem RKI ermöglichte es weltweit erstmalig, die epidemiologischen Zusammenhänge noch während des Ausbruchsgeschehens innerhalb weniger Tage aufzuklären. Auf diese Weise konnte die Quelle schnell identifiziert und der Ausbruch beendet werden. In Zusammenarbeit mit dem NRZ wurde hierzu außerdem eine spezifische diagnostische PCR-Methode entwickelt und bestätigt, um weitere Ausbrüche schnell und effektiv erkennen und bekämpfen zu können. „Dieses Beispiel illustriert eindrucksvoll die Möglichkeiten der molekularen Epidemiologie im DZIF“, unterstreicht Chakraborty.
Laut DZIF-Sprecher Prof. Dr. Martin Krönke hat sich die schleichende Ausbreitung multiresistenter Erreger in Deutschland inzwischen zu einer Bedrohung unbekannten Ausmaßes entwickelt. Diese Entwicklung wird das Öffentliche Gesundheitswesen in Zukunft vor große Probleme stellen.